
| XII: Vom Protein zum Gen III (INHALT) |
 |
Genbanken |
Klonierungsvektoren |
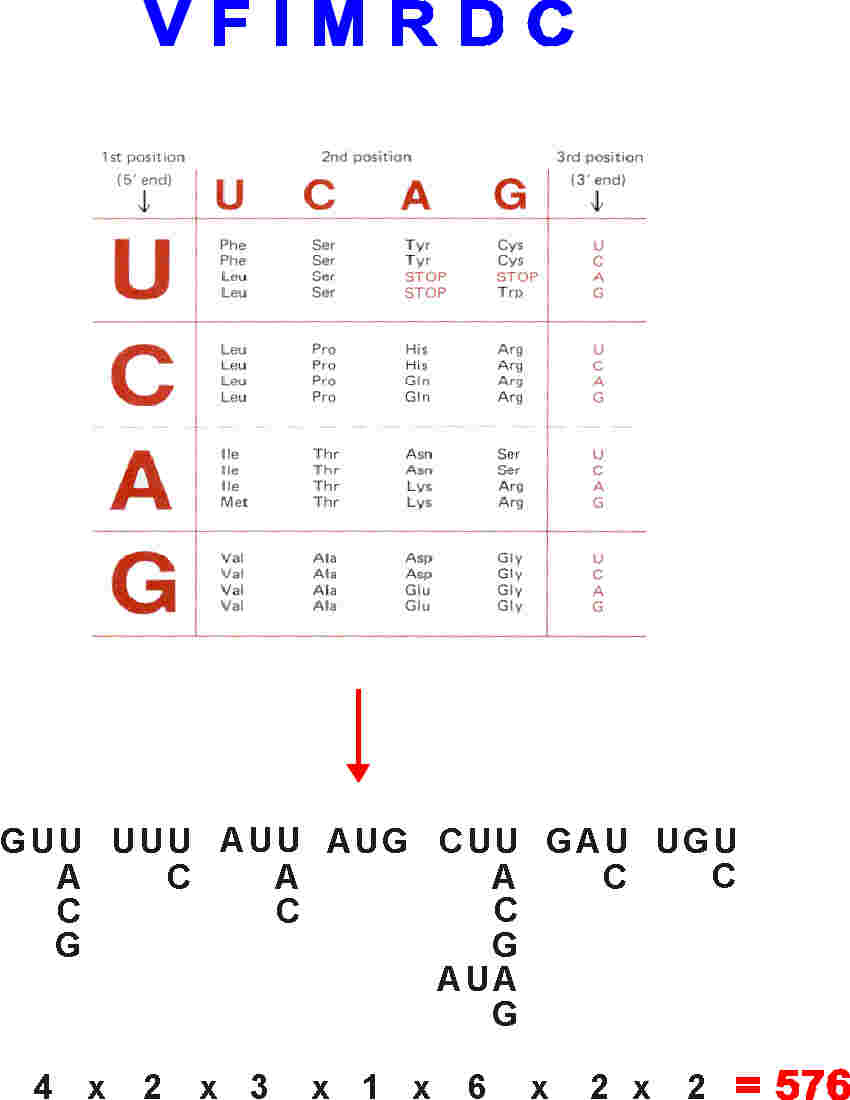
| Nachdem die Aminosäuresequenzen einiger Peptidfragmente des gereinigten Proteins bestimmt sind, werden diese Sequenzen in Nukleinsäuresequenzen übersetzt. In dem nebenstehenden Beispiel werden für jede Aminosäure alle Nukleinsäuretripletts aus der Tabelle herausgesucht und untereinander geschrieben. Für die gewählte Sequenz aus sieben Aminosäuren sind insgesamt 576 verschiedene Nukleinsäureabfolgen möglich. Dieses Sequenzgemisch (Oligonukleotide) wird in einem "Oligonukleotid-Synthesizer" chemisch hergestellt und später als Sonde zum Durchmustern von Genbibliotheken eingesetzt. |
|
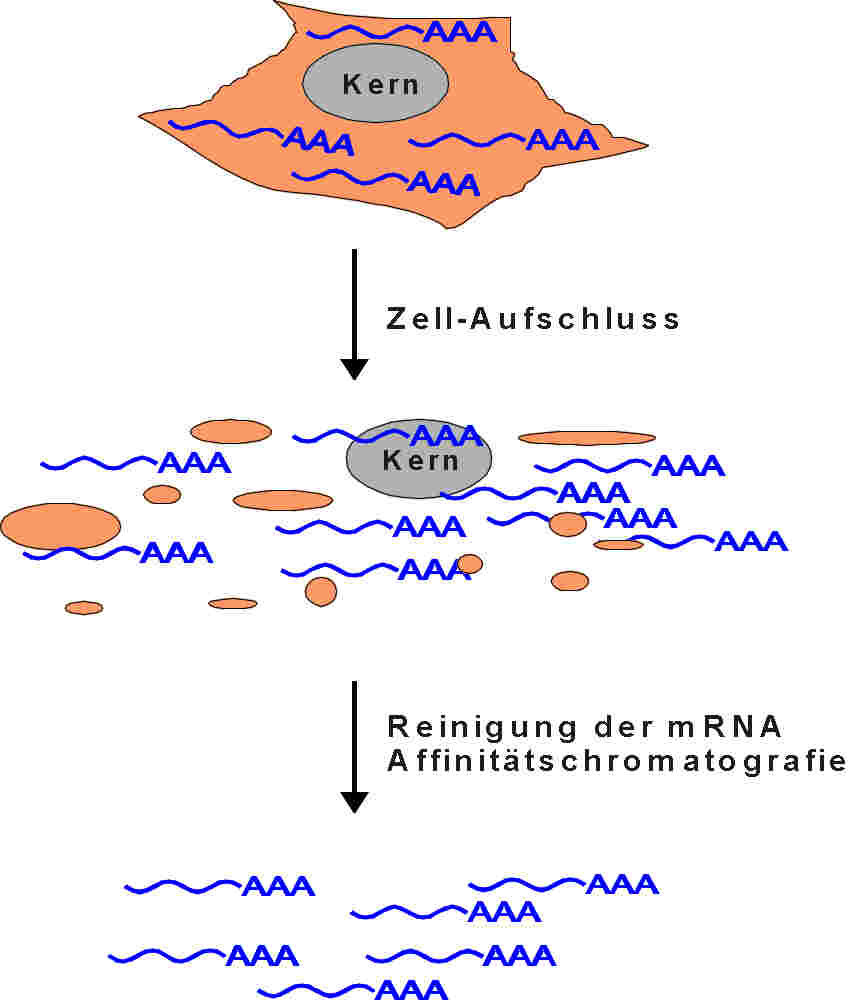
| Zur Reinigung des Proteins war Gewebe benutzt worden, das das Protein in großer Menge enthält. In erster Näherung kann davon ausgegangen werden, daß in dem Gewebe die Boten-RNA (mRNA), die für das Protein kodiert, ebenfalls in großer Menge vorkommt. Deshalb wird (in unserem Beispiel) das elektrische Organ des elektrischen Aals zur Isolierung der mRNA verwendet. Die Zellen werden aufgeschlossen und die mRNA über oligo(dT)-Zellulose affinitätschromatografisch gereinigt. |
|
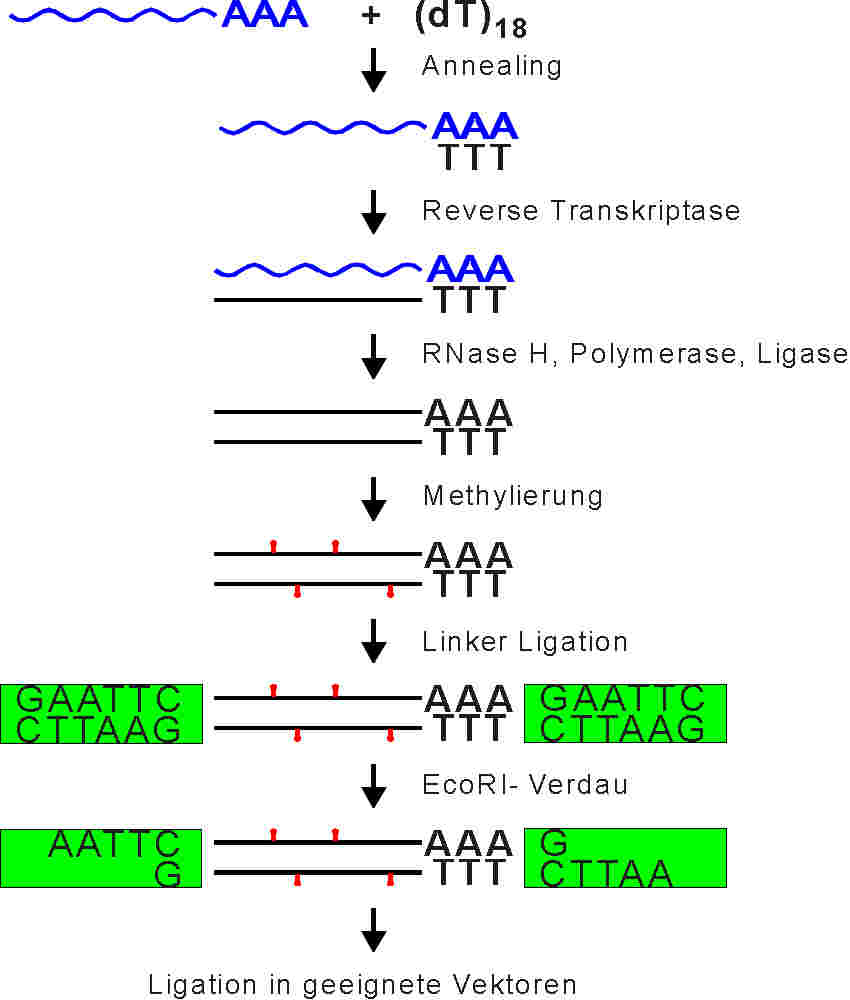
| Die isolierte mRNA wird in mehreren Schritten in doppelsträngige DNA (cDNA) umgeschrieben. Methylierung verhindert, daß die Moleküle bei der Inkubation mit Restriktionsenzymen gespalten werden. An die cDNA-Moleküle werden synthetisch hergestellte doppelsträngige DNA-Linker ligiert. Diese besitzen Erkennungssequenzen für bestimmte Restriktionsenzyme. Die Inkubation mit dem Restriktionsenzym (hier EcoRI) bereitet die cDNA-Moleküle für das Einfügen in geeignete Klonierungsvektoren vor. Als Vektoren für cDNA-Bibliotheken werden Bakteriophagen oder Plasmide verwendet. Gute cDNA-Bibliotheken enthalten mehrere 100.000 unabhängige Klone. |
|